資料介紹
描述
我們為什么要構建皮膚癌 AI?
根據皮膚癌基金會的數據,美國一半的人口在 65 歲之前被診斷出患有某種形式的皮膚癌。早期發現的存活率幾乎是 98%,但當癌癥到達淋巴結時,存活率會下降到 62%轉移到遠處器官時為 18%。借助皮膚癌 AI,我們希望利用人工智能的力量提供盡可能廣泛的早期檢測。
什么是人工智能以及如何使用它?
深度學習最近是機器學習的一大趨勢,最近的成功為構建這樣的項目鋪平了道路。在這個示例中,我們將特別關注計算機視覺和圖像分類。為此,我們將使用深度學習算法、卷積神經網絡 (CNN) 通過 Caffe 框架構建痣、黑色素瘤和脂溢性角化病圖像分類器。
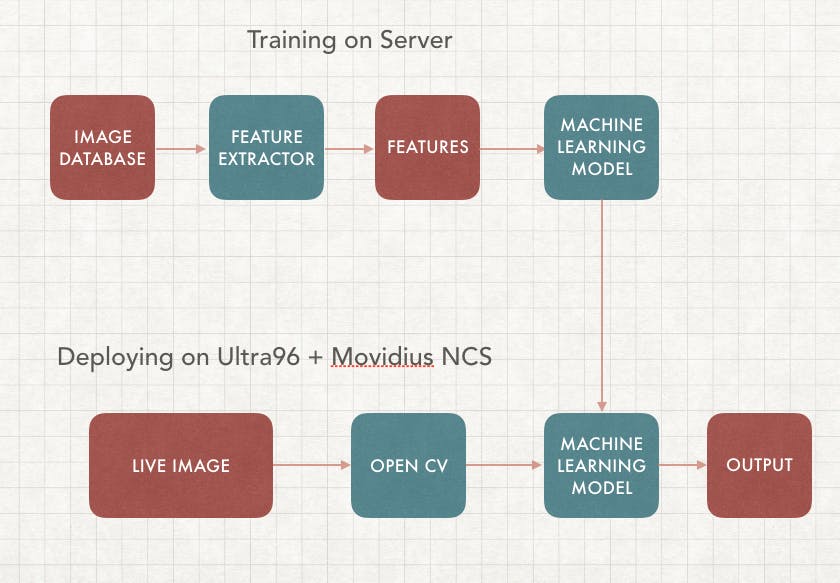
在本文中,我們將重點關注監督學習,它需要在服務器上進行訓練以及在邊緣部署。我們的目標是構建一個可以實時檢測癌癥圖像的機器學習算法,這樣您就可以構建自己的基于人工智能的皮膚癌分類設備。
我們的應用程序將包括兩部分,第一部分是訓練,我們將使用不同的癌癥圖像數據庫集來訓練具有相應標簽的機器學習算法(模型)。第二部分是在邊緣部署,它使用我們訓練過的相同模型并在邊緣設備上運行,在本例中是通過 Ultra96 FPGA 的 Movidius 神經計算棒。這樣 VPU 可以運行推理,而 FPGA 可以執行 OpenCV
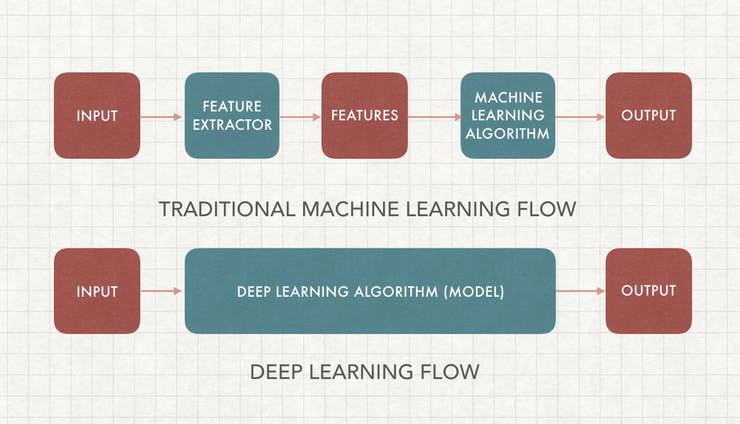
傳統機器學習 (ML) 與深度學習
這可能是人工智能中被問得最多的問題,一旦你學會了如何去做,它就相當簡單了。為了理解這一點,我們首先必須了解機器學習圖像分類的工作原理。
機器學習需要特征提取和模型訓練。我們首先必須使用領域知識來提取可用于我們的 ML 算法模型的特征,一些例子包括SIFT和HoG。之后,我們可以使用包含所有圖像特征和標簽的數據集來訓練我們的機器學習模型。
傳統 ML 和深度學習之間的主要區別在于特征工程。傳統 ML 使用手動編程的功能,而深度學習會自動執行。特征工程相對困難,因為它需要領域專業知識并且非常耗時。深度學習不需要特征工程,可以更準確
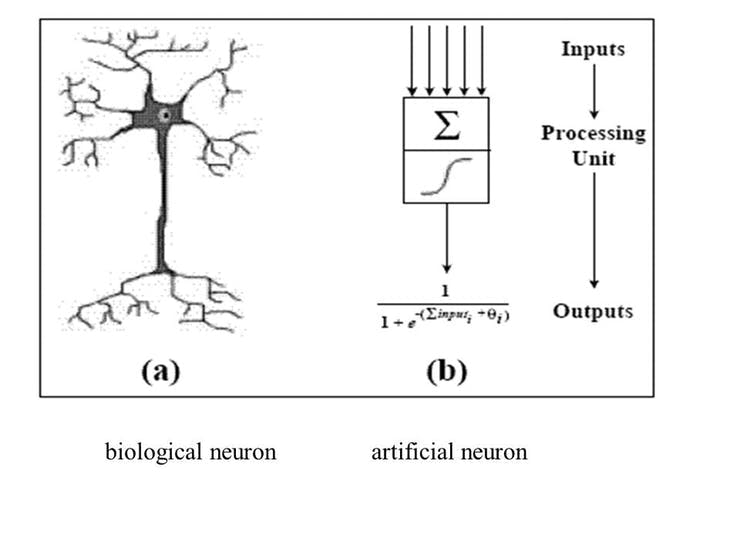
人工神經網絡 (ANN)
根據技術百科,“人工神經元網絡(ANN)是一種基于生物神經網絡結構和功能的計算模型。” 人工神經元網絡在技術上模擬人類生物神經元的工作方式,它具有有限數量的輸入、與它們相關的權重和激活功能。節點的激活函數定義了給定輸入或一組輸入的該節點的輸出,編碼復雜是非線性的。數據的模式。當輸入進來時,激活函數應用到輸入的權重和以生成輸出。人工神經元相互連接形成一個網絡,因此稱為人工神經元網絡(ANN)。
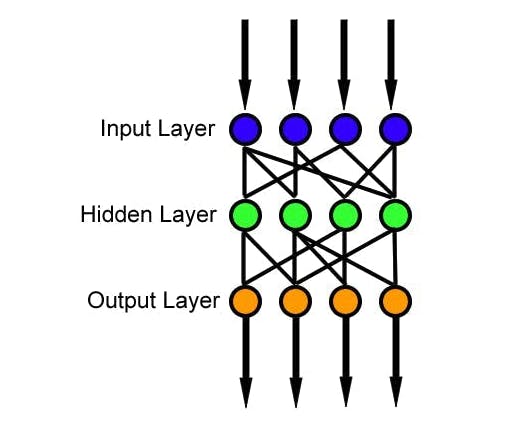
前饋神經網絡是一種人工神經網絡,其中節點之間的連接不形成循環,這是最簡單的人工神經網絡形式。它有 3 層,輸入層、隱藏層和輸出層,其中數據通過輸入層進入,通過隱藏層到達輸出節點,如下圖所示。我們可以有多個隱藏層,模型的復雜度與隱藏層的大小相關。
訓練數據和損失函數是用于訓練神經網絡的兩個元素。訓練數據由圖像和相應的標簽組成;損失函數是衡量分類過程中不準確的函數。一旦獲得了這兩個元素,我們就使用反向傳播算法和梯度下降來訓練 ANN。
卷積神經網絡 (CNN)
卷積神經網絡是一類深度前饋人工神經網絡,最常用于分析視覺圖像,因為它旨在模擬動物視覺皮層上的生物行為。它由卷積層和池化層組成,因此網絡可以對圖像屬性進行編碼。
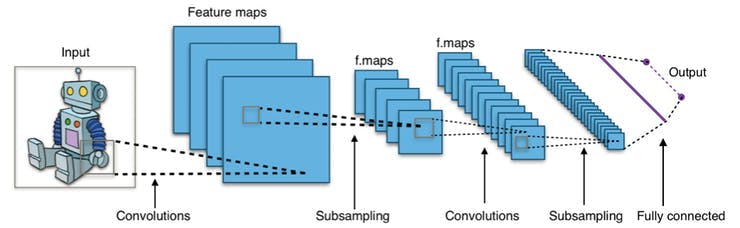
卷積層的參數由一組具有小感受野的可學習過濾器(或內核)組成。這樣,圖像可以在空間上進行卷積,計算過濾器條目和輸入之間的點積,并生成該過濾器的二維激活圖。通過這種方式,網絡可以學習在檢測到輸入圖像空間特征上的特殊特征時可以激活的過濾器。
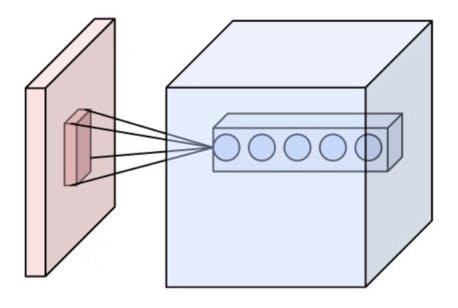
池化層是非線性下采樣的一種形式。它將輸入圖像劃分為一組不重疊的矩形,并為每個這樣的子區域輸出最大值。其思想是不斷減小輸入表示的空間大小以減少網絡中的參數量和計算量,因此它也可以控制過擬合。最大池化是最常見的非線性池化類型。根據維基百科,“池化通常與大小為 2x2 的過濾器一起應用,每個深度切片的步長為 2。大小為 2x2 且步長為 2 的池化層將輸入圖像縮小到其原始大小的 1/4。”
皮膚癌 AI 組件
該項目所需的設備非常簡單,您可以使用計算機和 USB Movidius 神經計算棒和 Ultra96 板來完成。
- Ultra96 板
- 內窺鏡相機
- Movidius 神經計算棒
- 屏幕或監視器

第 1 步:安裝 PYNQ Linux
Ultra96 是相當新的,但支持小組非常友好地讓基本的 Ubuntu 運行,這很重要,因為這讓我可以在 ultra96 上構建不同的平臺。編譯好的debian可以從https://fileserver.linaro.org/owncloud/index.php/s/jTt3MYSuwtLuf9d下載,之后我們可以使用ether之類的工具把它加載到mini-sd卡上。
啟動后,我們首先必須通過刪除損壞的存儲庫來修復一些錯誤。
sudo rm -r /var/lib/apt/lists/*
這使我們可以根據需要安裝所有軟件包以使用該平臺。
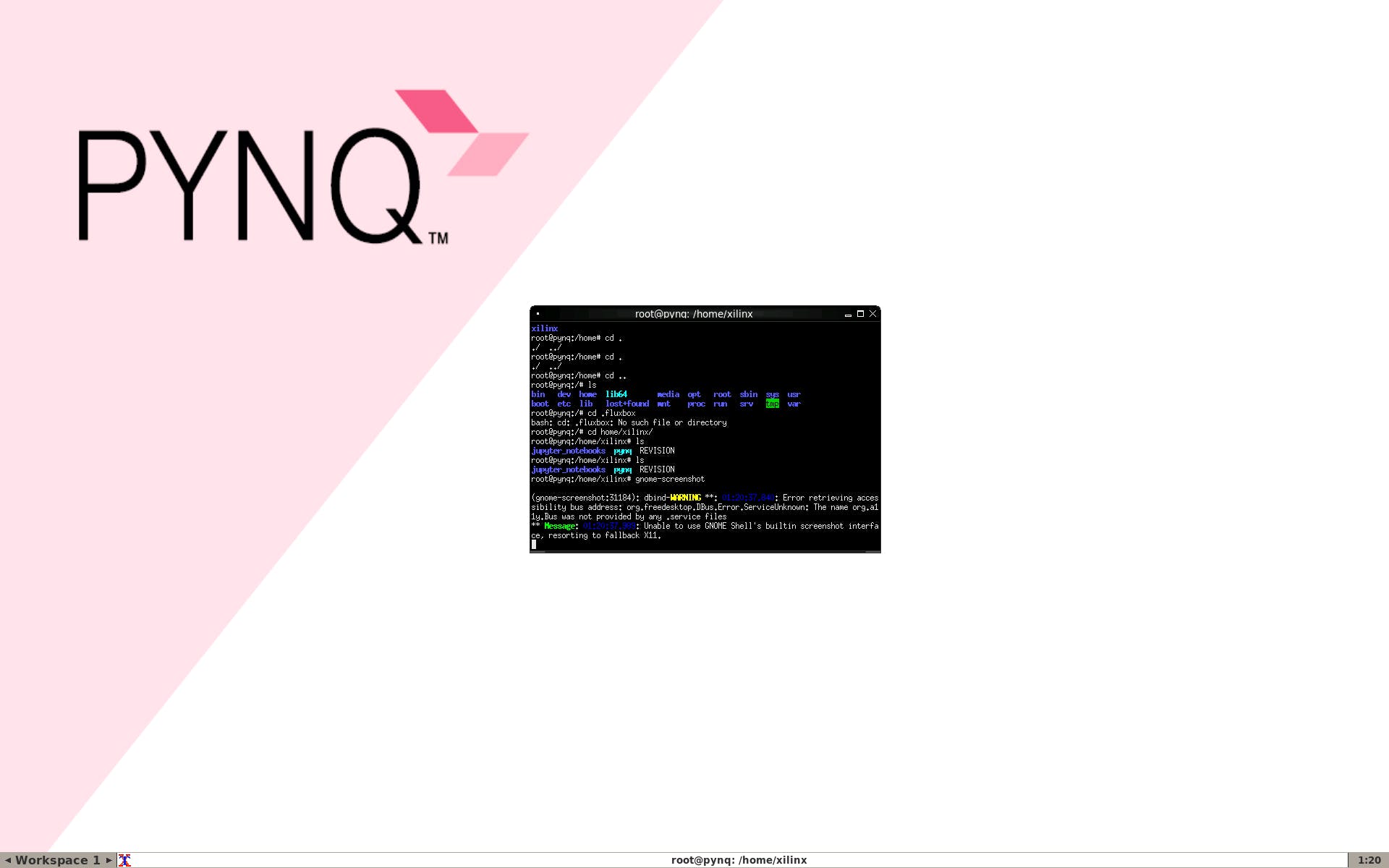
第 2 步:安裝 Movidius NCSDK
因此,為了讓 AI 和計算機視覺發揮作用,我們可以利用 Movidius NCS 來運行我們的項目。Ultra96 沒有演練,所以我們基本上通過 https://movidius.github.io/blog/ncs-apps-on-rpi/ 引用了這個
首先我們必須安裝依賴項,這部分不附帶 PYNQ,所以我們將安裝它們以確保一切正常。
apt-get install libgstreamer1.0-0 gstreamer1.0-plugins-base gstreamer1.0-plugins-good gstreamer1.0-plugins-bad gstreamer1.0-plugins-ugly gstreamer1.0-libav gstreamer1.0-doc gstreamer1.0-tools libgstreamer-plugins-base1.0-dev
apt-get install libgtk-3-dev
apt-get install -y libprotobuf-dev libleveldb-dev libsnappy-dev
apt-get install -y libopencv-dev libhdf5-serial-dev
apt-get install -y protobuf-compiler byacc libgflags-dev
apt-get install -y libgoogle-glog-dev liblmdb-dev libxslt-dev
接下來我們可以安裝 NCSDK SDK,它包含可以將應用程序連接到 NCS 的 API。由于 NCSDK 不是為 Ultra96 構建的,我們可以對markjay4k版本的解決方法進行以下修改
cd /home/xilinx/
mkdir -p workspace
cd workspace
git clone https://github.com/markjay4k/ncsdk-aarch64.git
cd ncsdk/api/src
make
make install
這個解決方法應該讓我們使用 Ultra96 和 PYNQ 的 NCSDK API
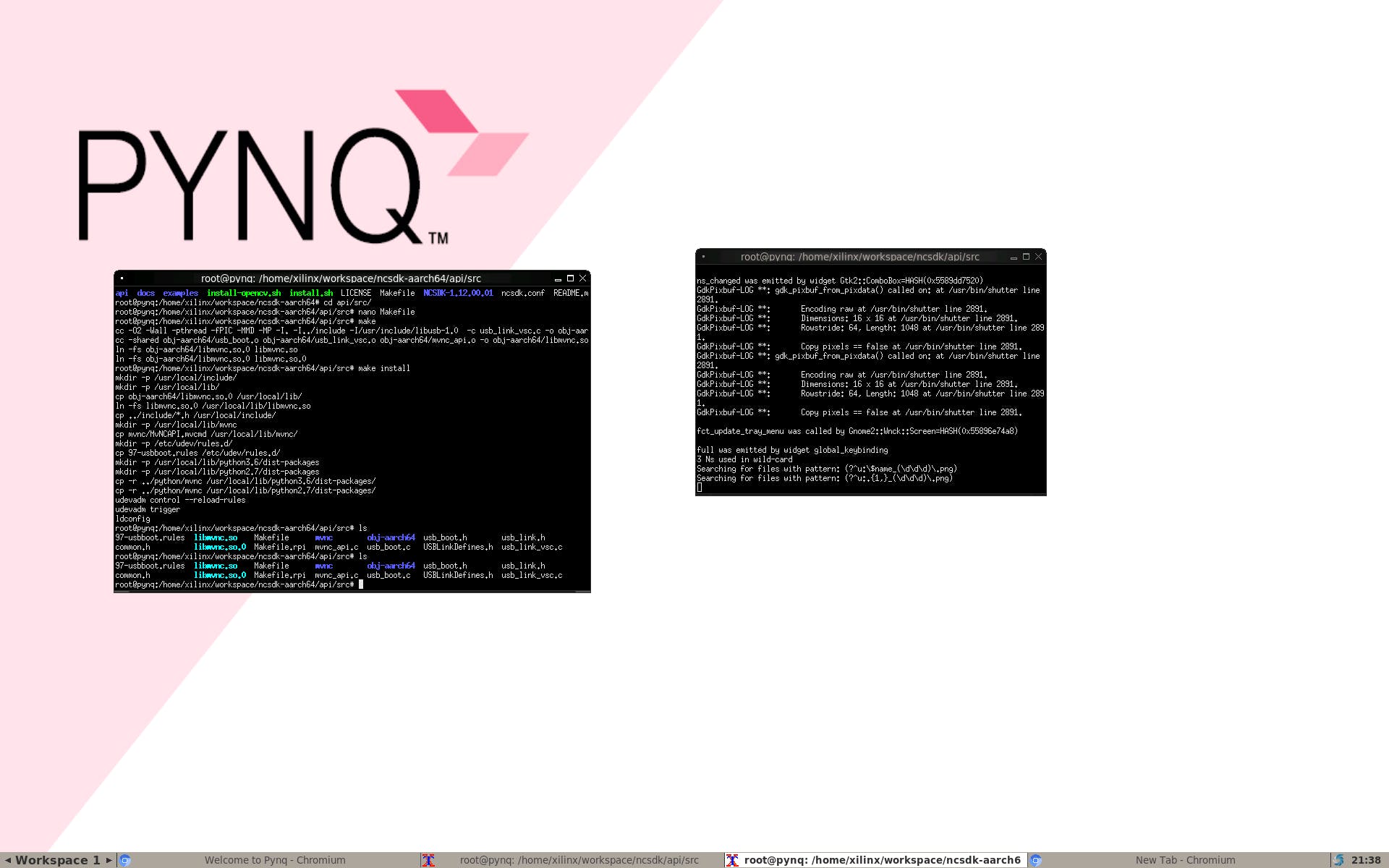
接下來我們將嘗試讓 NcAppZoo 和 Hello World 為 Neural Computing Stick 運行,我們需要在 pynq 上重復舊的 python 3.6
cd /home/xilinx/workspace
git clone https://github.com/movidius/ncappzoo
cd ncappzoo/apps/hello_ncs_py
make run
你剛剛讓 NCS 在 aarch64 上運行 :)
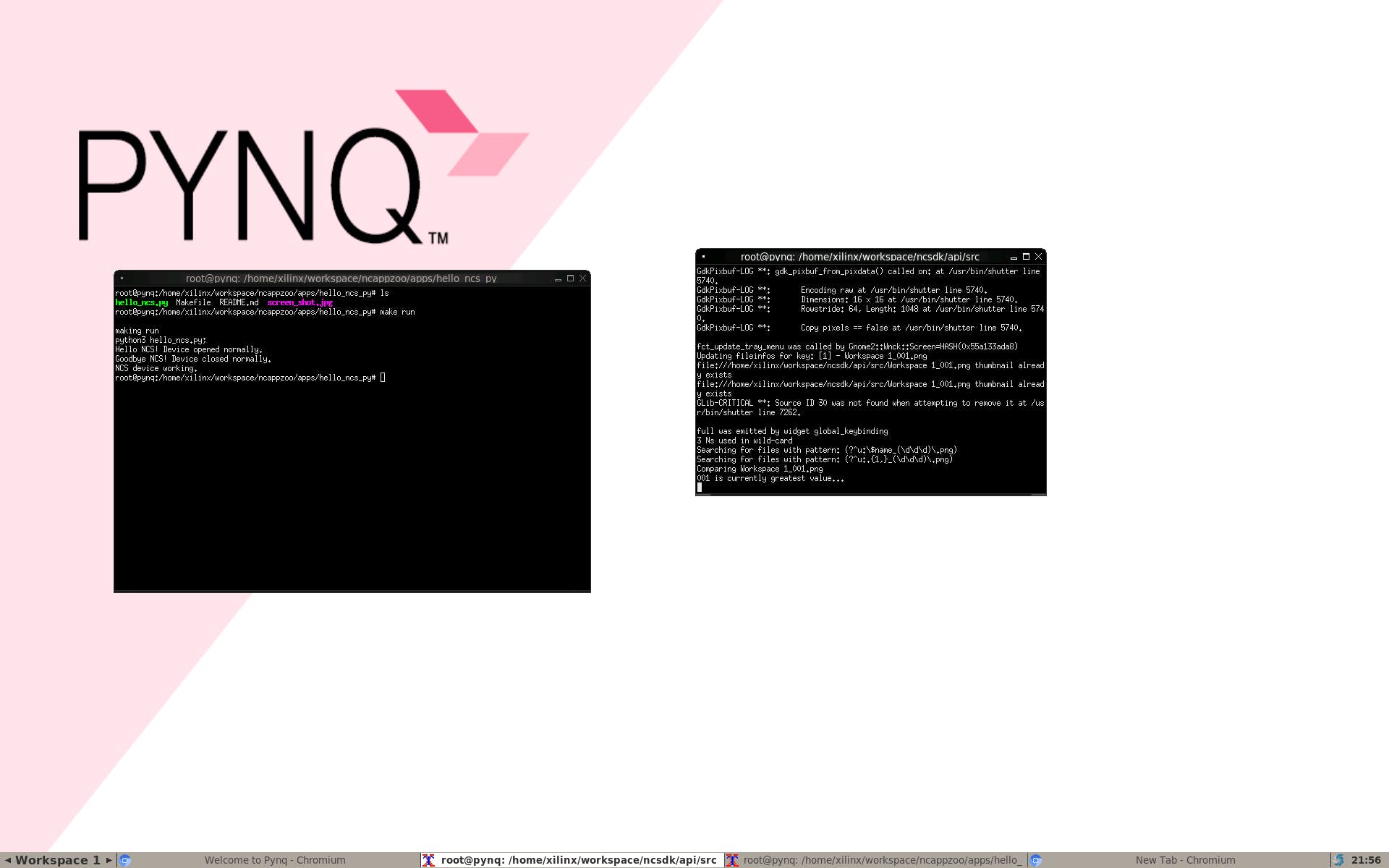
第 3 步:使用 Ultra96 和 NCS 實現邊緣人工智能
我們可以通過多種方式制作觸發器,因為這是一個基于 AI 的應用程序,我將制作一個基于 AI 的觸發器。在本指南中,我們將使用經過預訓練并使用 Caffe 的 SSD 神經網絡,檢測結果將是垃圾。因此,我們還將學習如何通過一些工作來利用其他神經網絡。

在這一步中,我們之前已經訓練了通過 caffe 模型,我們必須在另一臺機器上編譯圖形,因為我們只安裝了 API 而不是工具包,因為它們不是為 aarch64 構建的。然而,由于 API 有效,我們可以簡單地在另一臺機器上構建它并將圖形文件傳輸到 Ultra96。FPGA 可以處理所有的 CV2,這里的 Movidius NCS 將運行推理以進行對象檢測和圖像分類,如下圖所示,我們將通過在這里進行實時圖像分類來確定基礎。
第 4 步:收集皮膚癌的圖像數據集
我們首先需要皮膚癌數據集,雖然有很多地方可以獲取,但 isic-archive 成為最簡單的一個。對于這部分,我們只需要大約 500 張痣、黑色素瘤和脂溢性角化病之間的圖像,以及 500 張其他任何東西的隨機圖像。為了獲得更高的準確性,我們必須使用更多的數據,這只是為了開始訓練。獲取數據的最簡單方法是通過下面的圖片使用 https://isic-archive.com/#images
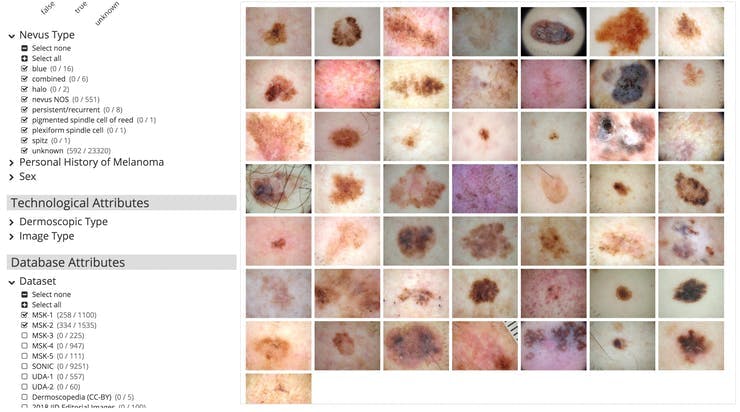
之后,我們可以將圖像標記到一個文件夾中,并將它們命名為 nevus-00.jpg、melanoma-00.jpg 以便我們可以輕松地構建我們的 lmdb。
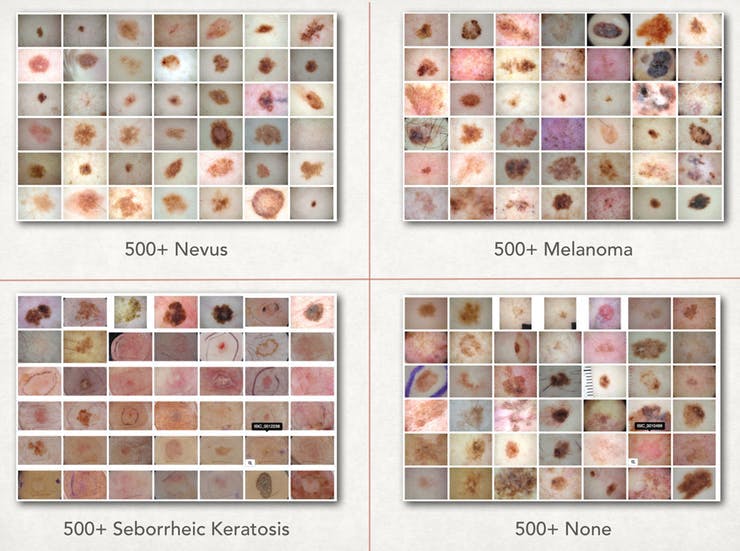
第 5 步:設置訓練服務器
機器學習訓練使用了大量的處理能力,因此它們通常成本很高。在本文中,我們將重點介紹面向英特爾 AI Academy成員免費提供的 AI DevCloud。
為了構建皮膚癌 AI,我們使用 Caffe 框架,主要是因為英特爾 Movidius NCS 的 AI on the Edge 支持。Caffe 是由伯克利視覺與學習中心 (BVLC) 開發的深度學習框架,使用 caffe 框架訓練我們的算法模型有 4 個步驟。這樣,Xilinx 的 FPGA 可以專注于 openCV,NCS 可以專注于推理。
- 數據準備,我們清理圖像并將它們存儲到 LMDB。
- 模型定義 prototxt 文件,定義參數并選擇 CNN 架構。
- Solver Definition prototxt 文件,定義模型優化的求解器參數
- 模型訓練,我們執行caffe命令得到我們的.caffemodel算法文件
在 Devcloud 上,我們可以檢查這些是否可用,只需轉到
cd /glob/deep-learning/py-faster-rcnn/caffe-fast-rcnn/build/tools
第 6 步:準備 LMDB 進行訓練
一旦我們設置好 DevCloud,我們就可以構建它
mkdir skincancerai
cd skincancerai
mkdir input
cd input
mkdir train
從那里我們可以將我們之前設置的所有數據放入文件夾中
scp ./* colfax:/home/[youruser_name]/skincancerai/input/train/
之后,我們可以通過這種方式構建我們的 lmdb。我們通過以下幾組來做到這一點
- 數據集的 5/6 將用于訓練,1/6 用于驗證,因此我們可以計算模型的準確性
- 我們將所有圖像的大小調整為 227x227,以遵循與 BVLC 相同的標準
- 直方圖均衡化應用于所有訓練圖像以調整對比度。
- 并將它們存儲在 train_lmdb 和 validation_lmdb 中
- 使用 make_datum 標記 lmdb 內的所有圖像數據集
import os
import glob
import random
import numpy as np
import cv2
import caffe
from caffe.proto import caffe_pb2
import lmdb
#We use 227x227 from BVLC
IMAGE_WIDTH = 227
IMAGE_HEIGHT = 227
def transform_img(img, img_width=IMAGE_WIDTH, img_height=IMAGE_HEIGHT):
img[:, :, 0] = cv2.equalizeHist(img[:, :, 0])
img[:, :, 1] = cv2.equalizeHist(img[:, :, 1])
img[:, :, 2] = cv2.equalizeHist(img[:, :, 2])
img = cv2.resize(img, (img_width, img_height), interpolation = cv2.INTER_CUBIC)
return img
def make_datum(img, label):
return caffe_pb2.Datum(
channels=3,
width=IMAGE_WIDTH,
height=IMAGE_HEIGHT,
label=label,
data=np.rollaxis(img, 2).tostring())
train_lmdb = '/home/[your_username]/skincancerai/input/train_lmdb'
validation_lmdb = '/home/[youser_username]/skincancerai/input/validation_lmdb'
os.system('rm -rf ' + train_lmdb)
os.system('rm -rf ' + validation_lmdb)
train_data = [img for img in glob.glob("./input/train/*jpg")]
random.shuffle(train_data)
print 'Creating train_lmdb'
in_db = lmdb.open(train_lmdb, map_size=int(1e12))
with in_db.begin(write=True) as in_txn:
for in_idx, img_path in enumerate(train_data):
if in_idx % 6 == 0:
continue
img = cv2.imread(img_path, cv2.IMREAD_COLOR)
img = transform_img(img, img_width=IMAGE_WIDTH, img_height=IMAGE_HEIGHT)
if 'none' in img_path:
label = 0
elif 'nevus' in img_path:
label = 1
elif 'melanoma' in img_path:
label = 2
else:
label = 3
datum = make_datum(img, label)
in_txn.put('{:0>5d}'.format(in_idx), datum.SerializeToString())
print '{:0>5d}'.format(in_idx) + ':' + img_path
in_db.close()
print '\nCreating validation_lmdb'
in_db = lmdb.open(validation_lmdb, map_size=int(1e12))
with in_db.begin(write=True) as in_txn:
for in_idx, img_path in enumerate(train_data):
if in_idx % 6 != 0:
continue
img = cv2.imread(img_path, cv2.IMREAD_COLOR)
img = transform_img(img, img_width=IMAGE_WIDTH, img_height=IMAGE_HEIGHT)
if 'none' in img_path:
label = 0
elif 'nevus' in img_path:
label = 1
elif 'melanoma' in img_path:
label = 2
else:
label = 3
datum = make_datum(img, label)
in_txn.put('{:0>5d}'.format(in_idx), datum.SerializeToString())
print '{:0>5d}'.format(in_idx) + ':' + img_path
in_db.close()
print '\nFinished processing all images'
之后我們將運行腳本
python2 create_lmdb.py
獲取所有 LMDB。完成后,我們需要獲取訓練數據的平均圖像。作為 caffe 的一部分,我們可以通過
cd /glob/deep-learning/py-faster-rcnn/caffe-fast-rcnn/build/toolscompute_image_mean -backend=lmdb /home/[your_user]/skincancerai/input/train_lmdb /home/[your_user]/skincancerai/input/mean.binaryproto
上面的命令將生成訓練數據的平均圖像。每個輸入圖像將減去平均圖像,使每個特征像素的均值為零。這是監督機器學習預處理中常用的方法。
第 7 步:設置模型定義和求解器定義
我們現在需要設置模型定義和求解器定義,在本文中我們將使用 bvlc_reference_net,可以在https://github.com/BVLC/caffe/tree/master/models/bvlc_reference_caffenet看到
下面是 train.prototxt 的修改版本
name: "CaffeNet"
layer {
name: "data"
type: "Data"
top: "data"
top: "label"
include {
phase: TRAIN
}
transform_param {
mirror: true
crop_size: 227
mean_file: "/home/[your_username]/skincancerai/input/mean.binaryproto"
}
data_param {
source: "/home/[your_username]/skincancerai/input/train_lmdb"
batch_size: 128
backend: LMDB
}
}
layer {
name: "data"
type: "Data"
top: "data"
top: "label"
include {
phase: TEST
}
transform_param {
mirror: false
crop_size: 227
mean_file: "/home/[your_username]/skincancerai/input/mean.binaryproto"
}
# mean pixel / channel-wise mean instead of mean image
# transform_param {
# crop_size: 227
# mean_value: 104
# mean_value: 117
# mean_value: 123
# mirror: true
# }
data_param {
source: "/home/[your_username]/skincancerai/input/validation_lmdb"
batch_size: 36
backend: LMDB
}
}
layer {
name: "conv1"
type: "Convolution"
bottom: "data"
top: "conv1"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
convolution_param {
num_output: 96
kernel_size: 11
stride: 4
weight_filler {
type: "gaussian"
std: 0.01
}
bias_filler {
type: "constant"
value: 0
}
}
}
layer {
name: "relu1"
type: "ReLU"
bottom: "conv1"
top: "conv1"
}
layer {
name: "pool1"
type: "Pooling"
bottom: "conv1"
top: "pool1"
pooling_param {
pool: MAX
kernel_size: 3
stride: 2
}
}
layer {
name: "norm1"
type: "LRN"
bottom: "pool1"
top: "norm1"
lrn_param {
local_size: 5
alpha: 0.0001
beta: 0.75
}
}
layer {
name: "conv2"
type: "Convolution"
bottom: "norm1"
top: "conv2"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
convolution_param {
num_output: 256
pad: 2
kernel_size: 5
group: 2
weight_filler {
type: "gaussian"
std: 0.01
}
bias_filler {
type: "constant"
value: 1
}
}
}
layer {
name: "relu2"
type: "ReLU"
bottom: "conv2"
top: "conv2"
}
layer {
name: "pool2"
type: "Pooling"
bottom: "conv2"
top: "pool2"
pooling_param {
pool: MAX
kernel_size: 3
stride: 2
}
}
layer {
name: "norm2"
type: "LRN"
bottom: "pool2"
top: "norm2"
lrn_param {
local_size: 5
alpha: 0.0001
beta: 0.75
}
}
layer {
name: "conv3"
type: "Convolution"
bottom: "norm2"
top: "conv3"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
convolution_param {
num_output: 384
pad: 1
kernel_size: 3
weight_filler {
type: "gaussian"
std: 0.01
}
bias_filler {
type: "constant"
value: 0
}
}
}
layer {
name: "relu3"
type: "ReLU"
bottom: "conv3"
top: "conv3"
}
layer {
name: "conv4"
type: "Convolution"
bottom: "conv3"
top: "conv4"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
convolution_param {
num_output: 384
pad: 1
kernel_size: 3
group: 2
weight_filler {
type: "gaussian"
std: 0.01
}
bias_filler {
type: "constant"
value: 1
}
}
}
layer {
name: "relu4"
type: "ReLU"
bottom: "conv4"
top: "conv4"
}
layer {
name: "conv5"
type: "Convolution"
bottom: "conv4"
top: "conv5"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
convolution_param {
num_output: 256
pad: 1
kernel_size: 3
group: 2
weight_filler {
type: "gaussian"
std: 0.01
}
bias_filler {
type: "constant"
value: 1
}
}
}
layer {
name: "relu5"
type: "ReLU"
bottom: "conv5"
top: "conv5"
}
layer {
name: "pool5"
type: "Pooling"
bottom: "conv5"
top: "pool5"
pooling_param {
pool: MAX
kernel_size: 3
stride: 2
}
}
layer {
name: "fc6"
type: "InnerProduct"
bottom: "pool5"
top: "fc6"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
inner_product_param {
num_output: 4096
weight_filler {
type: "gaussian"
std: 0.005
}
bias_filler {
type: "constant"
value: 1
}
}
}
layer {
name: "relu6"
type: "ReLU"
bottom: "fc6"
top: "fc6"
}
layer {
name: "drop6"
type: "Dropout"
bottom: "fc6"
top: "fc6"
dropout_param {
dropout_ratio: 0.5
}
}
layer {
name: "fc7"
type: "InnerProduct"
bottom: "fc6"
top: "fc7"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
inner_product_param {
num_output: 4096
weight_filler {
type: "gaussian"
std: 0.005
}
bias_filler {
type: "constant"
value: 1
}
}
}
layer {
name: "relu7"
type: "ReLU"
bottom: "fc7"
top: "fc7"
}
layer {
name: "drop7"
type: "Dropout"
bottom: "fc7"
top: "fc7"
dropout_param {
dropout_ratio: 0.5
}
}
layer {
name: "fc8"
type: "InnerProduct"
bottom: "fc7"
top: "fc8"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
inner_product_param {
num_output: 4
weight_filler {
type: "gaussian"
std: 0.01
}
bias_filler {
type: "constant"
value: 0
}
}
}
layer {
name: "accuracy"
type: "Accuracy"
bottom: "fc8"
bottom: "label"
top: "accuracy"
include {
phase: TEST
}
}
layer {
name: "loss"
type: "SoftmaxWithLoss"
bottom: "fc8"
bottom: "label"
top: "loss"
}

同時我們可以創建基于 train.prototxt 構建的 deploy.prototxt。這可以從 github repo 中看到。我們還將像創建 lmdb 文件一樣創建 label.txt 文件
classes
None
Nevus
Melanoma
Seborrheic Keratosis
之后我們需要solver.prototxt中的Solver Definition,它用于優化訓練模型。因為我們依賴CPU,所以我們需要對下面的求解器定義做一些修改。
net: "/home/[your_username]/skincancerai/model/train.prototxt"
test_iter: 50
test_interval: 50
base_lr: 0.001
lr_policy: "step"
gamma: 0.1
stepsize: 50
display: 50
max_iter: 5000
momentum: 0.9
weight_decay: 0.0005
snapshot: 1000
snapshot_prefix: "/home/[your_username]/skincancerai/model"
solver_mode: CPU
因為我們在這里處理的數據量很小,所以我們可以縮短測試迭代并盡可能快地獲得我們的模型。簡而言之,求解器將使用驗證集每 50 次迭代計算模型的準確性。由于我們沒有大量數據,求解器優化過程將每 1000 次迭代拍攝一次快照,最多運行 5000 次迭代。lr_policy: "step", stepsize: 2500, base_lr: 0.001 and gamma: 0.1 的當前配置是相當標準的,因為我們也可以通過bvlc 求解器文檔嘗試使用其他配置。
第 8 步:訓練模型
由于我們使用的是免費的 AI DevCloud 并且一切就緒,我們可以使用Intel Caffe ,它使用安裝在集群上的 Intel CPU 進行了優化。由于這是一個集群,我們可以使用以下命令簡單地開始訓練。
cd /glob/deep-learning/py-faster-rcnn/caffe-fast-rcnn/build/toolsecho caffe train --solver ~/skincancerai/model/solver.prototxt | qsub -o ~/skincancerai/model/output.txt -e ~/skincancerai/model/train.log
訓練后的模型將是model_iter_1000.caffemodel、model_iter_2000.caffemodel等。使用 ISIC 提供的數據,您應該獲得大約 70% 到 80% 的準確度。您可以通過以下命令繪制自己的曲線,
cd ~/skincancerai python2 plot_learning_curve.py ./model/train.log ./model/train.png
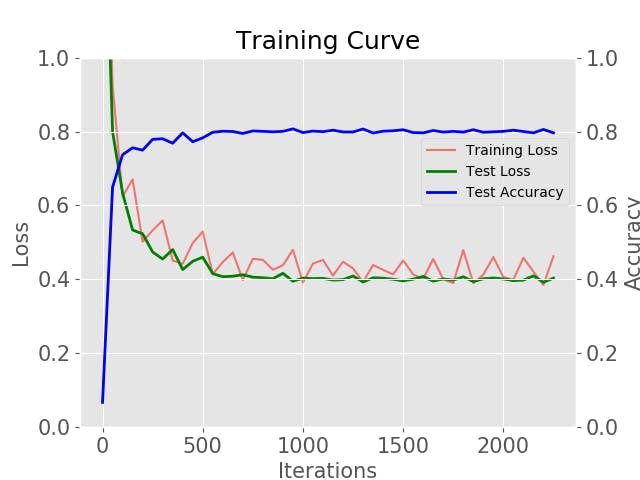
第 9 步:在具有 NCS 的 Ultra96 上部署和運行
在本文中,我們使用 Ultra96,這樣我們就可以擁有一個可以隨身攜帶的離線設備。我們已經在步驟 3 上運行了示例。由于我們在 aarch64 上沒有激活工具包,我們可以在另一臺機器上運行編譯,然后將其復制到 ultra96
mvNCCompile deploy.prototxt -w
model_iter_1000.caffemodel,model_iter_2000.caffemodel
這將為您提供圖形文件,復制圖形文件和標簽文件并將其與我們修改的以下代碼一起放在 CancerNet 下,將其復制到 Ultra96。我們現在有一個可以在家中使用的皮膚癌檢測設備。我們可以在CancerNet下使用圖形文件和categories.txt文件部署以下代碼
#!/usr/bin/python3
# ****************************************************************************
# Copyright(c) 2017 Intel Corporation.
# License: MIT See LICENSE file in root directory.
# ****************************************************************************
# Perform inference on a LIVE camera feed using DNNs on
# Intel? Movidius? Neural Compute Stick (NCS)
import os
import cv2
import sys
import numpy
import ntpath
import argparse
import mvnc.mvncapi as mvnc
# Variable to store commandline arguments
ARGS = None
# OpenCV object for video capture
camera = None
# ---- Step 1: Open the enumerated device and get a handle to it -------------
def open_ncs_device():
# Look for enumerated NCS device(s); quit program if none found.
devices = mvnc.EnumerateDevices()
if len( devices ) == 0:
print( "No devices found" )
quit()
# Get a handle to the first enumerated device and open it
device = mvnc.Device( devices[0] )
device.OpenDevice()
return device
# ---- Step 2: Load a graph file onto the NCS device -------------------------
def load_graph( device ):
# Read the graph file into a buffer
with open( ARGS.graph, mode='rb' ) as f:
blob = f.read()
# Load the graph buffer into the NCS
graph = device.AllocateGraph( blob )
return graph
# ---- Step 3: Pre-process the images ----------------------------------------
def pre_process_image( frame ):
# Resize image [Image size is defined by choosen network, during training]
img = cv2.resize( frame, tuple( ARGS.dim ) )
# Extract/crop a section of the frame and resize it
height, width, channels = frame.shape
x1 = int( width / 3 )
y1 = int( height / 4 )
x2 = int( width * 2 / 3 )
y2 = int( height * 3 / 4 )
cv2.rectangle( frame, ( x1, y1 ) , ( x2, y2 ), ( 0, 255, 0 ), 2 )
img = frame[ y1 : y2, x1 : x2 ]
# Resize image [Image size if defined by choosen network, during training]
img = cv2.resize( img, tuple( ARGS.dim ) )
# Convert BGR to RGB [OpenCV reads image in BGR, some networks may need RGB]
if( ARGS.colormode == "rgb" ):
img = img[:, :, ::-1]
# Mean subtraction & scaling [A common technique used to center the data]
img = img.astype( numpy.float16 )
img = ( img - numpy.float16( ARGS.mean ) ) * ARGS.scale
return img
# ---- Step 4: Read & print inference results from the NCS -------------------
def infer_image( graph, img, frame ):
# Load the image as a half-precision floating point array
graph.LoadTensor( img, 'user object' )
# Get the results from NCS
output, userobj = graph.GetResult()
# Find the index of highest confidence
top_prediction = output.argmax()
# Get execution time
inference_time = graph.GetGraphOption( mvnc.GraphOption.TIME_TAKEN )
print( "I am %3.1f%%" % (100.0 * output[top_prediction] ) + " confidant"
+ " it is " + labels[top_prediction]
+ " ( %.2f ms )" % ( numpy.sum( inference_time ) ) )
displaystring = str(100.0 * output[top_prediction]) + " " + labels[top_prediction]
# If a display is available, show the image on which inference was performed
if 'DISPLAY' in os.environ:
textsize = cv2.getTextSize(displaystring, cv2.FONT_HERSHEY_SIMPLEX, 1, 2)[0]
textX = (frame.shape[1] - textsize[0])/2
cv2.putText(frame, displaystring, (int(textX),450), cv2.FONT_HERSHEY_SIMPLEX, 1, (255,0,0),2)
cv2.imshow( 'Skin Cancer AI', frame )
# ---- Step 5: Unload the graph and close the device -------------------------
def close_ncs_device( device, graph ):
graph.DeallocateGraph()
device.CloseDevice()
camera.release()
cv2.destroyAllWindows()
# ---- Main function (entry point for this script ) --------------------------
def main():
device = open_ncs_device()
graph = load_graph( device )
# Main loop: Capture live stream & send frames to NCS
while( True ):
ret, frame = camera.read()
img = pre_process_image( frame )
infer_image( graph, img, frame )
# Display the frame for 5ms, and close the window so that the next
# frame can be displayed. Close the window if 'q' or 'Q' is pressed.
if( cv2.waitKey( 5 ) & 0xFF == ord( 'q' ) ):
break
close_ncs_device( device, graph )
# ---- Define 'main' function as the entry point for this script -------------
if __name__ == '__main__':
parser = argparse.ArgumentParser(
description="Image classifier using \
Intel? Movidius? Neural Compute Stick." )
parser.add_argument( '-g', '--graph', type=str,
default='CancerNet/graph',
help="Absolute path to the neural network graph file." )
parser.add_argument( '-v', '--video', type=int,
default=0,
help="Index of your computer's V4L2 video device. \
ex. 0 for /dev/video0" )
parser.add_argument( '-l', '--labels', type=str,
default='./CancerNet/categories.txt',
help="Absolute path to labels file." )
parser.add_argument( '-M', '--mean', type=float,
nargs='+',
default=[78.42633776, 87.76891437, 114.89584775],
help="',' delimited floating point values for image mean." )
parser.add_argument( '-S', '--scale', type=float,
default=1,
help="Absolute path to labels file." )
parser.add_argument( '-D', '--dim', type=int,
nargs='+',
default=[227, 227],
help="Image dimensions. ex. -D 224 224" )
parser.add_argument( '-c', '--colormode', type=str,
default="rgb",
help="RGB vs BGR color sequence. This is network dependent." )
ARGS = parser.parse_args()
# Create a VideoCapture object
camera = cv2.VideoCapture( ARGS.video )
# Set camera resolution
camera.set( cv2.CAP_PROP_FRAME_WIDTH, 620 )
camera.set( cv2.CAP_PROP_FRAME_HEIGHT, 480 )
# Load the labels file
labels =[ line.rstrip('\n') for line in
open( ARGS.labels ) if line != 'classes\n']
main()
# ==== End of file ===========================================================
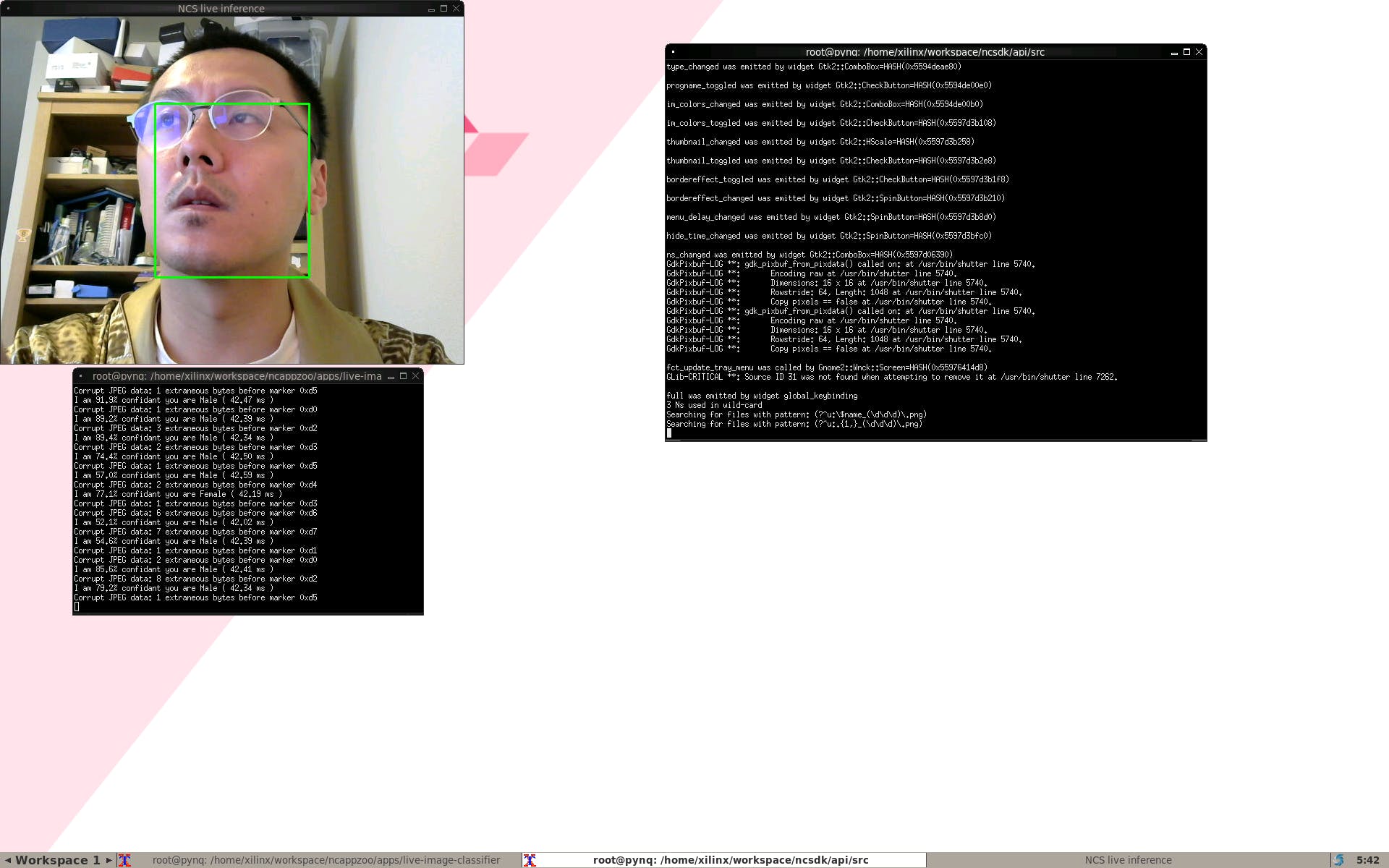
從這里開始,我們就有了皮膚癌 AI 分類,只需在文件夾中運行以下命令。
python3 live-image-classifier.py
我們可以檢測到我們自己的痣
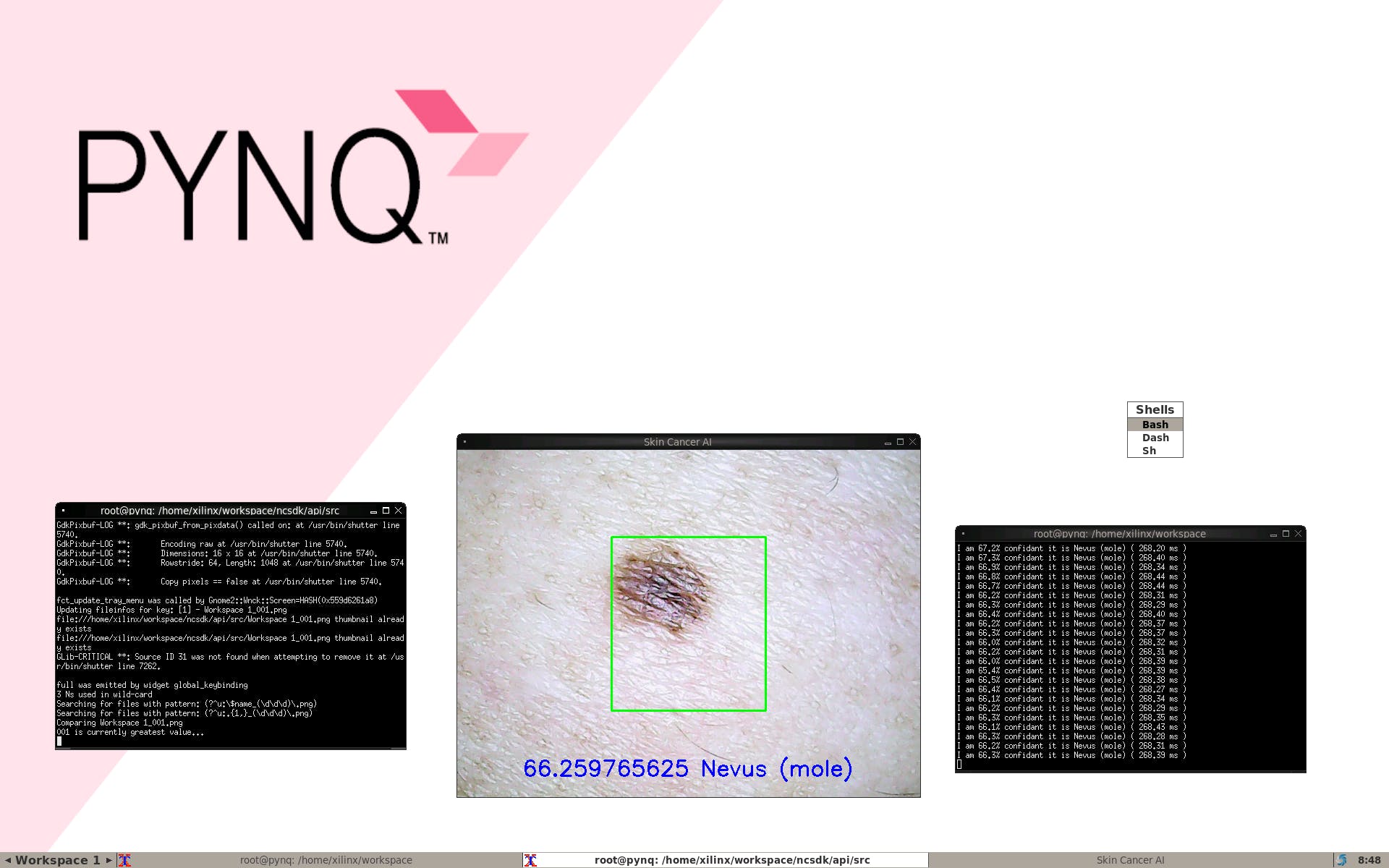
當有黑色素瘤時
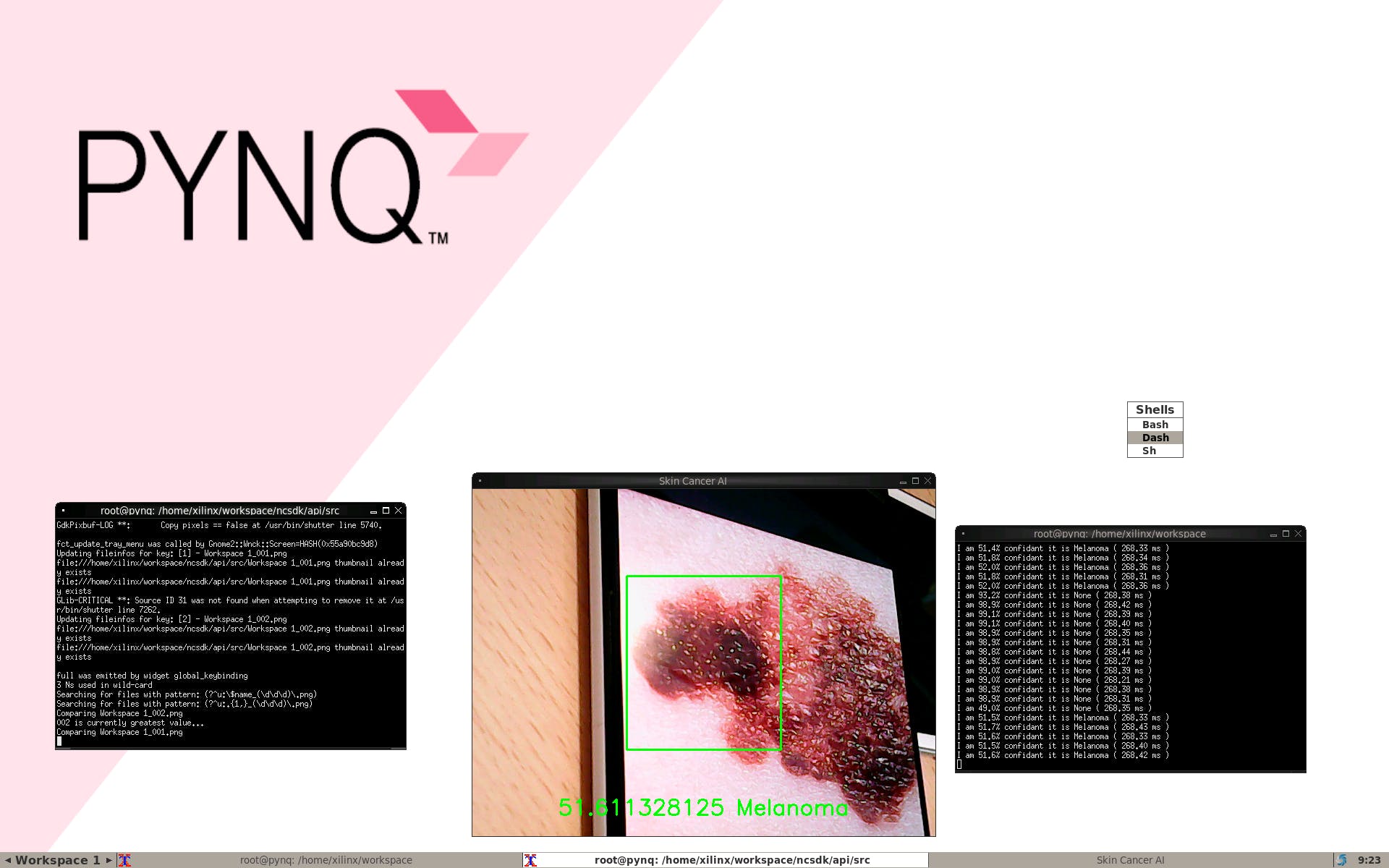
我們可以看到完整的演示
?
- Ultra96硬件用戶指南
- Ultra96 CSI-2視頻輸出到Raspberry Pi攝像頭輸入
- Ultra96上的實時攝像頭饋送網頁
- 使用PYNQ的Ultra96面部識別鎖栓
- 使用Tensil、TF-Lite和PYNQ在Ultra96板上運行YOLO v4 Tiny
- 在Ultra96 V2平臺上用Python實現人臉檢測和人臉跟蹤
- 用于Ultra96的夾層板96AnalogXperience
- Ultra96 FPGA上的Live NYC Subway Monitor應用程序
- 關于Ultra96的Xilinx DDS編譯器IP教程
- 與Ultra96聯網端口轉發
- Ultra96 V2上基于標記的增強現實
- 使用Ultra96 PYNQ測定織物GSM
- 2018.2 Ultra96:從 Matchbox 桌面關斷 PetaLinux BSP,無法關斷電路板
- 一起玩Ultra96之GPIO操作
- 紫外線監測UV指數傳感器讓人們免于暴曬防止皮膚癌 2次下載
- 在NVIDIA Holoscan SDK中使用OpenCV構建零拷貝AI傳感器處理管線 554次閱讀
- 全植入無線觸覺系統:仿生人造皮膚在傷口愈合與觸覺恢復中的應用 765次閱讀
- 電子皮膚是一項前沿技術,具有廣闊的應用前景 3380次閱讀
- 柔性電子皮膚具有神經形態感知-運動回路 1118次閱讀
- 基于AI視覺技術構建柔性生產數字化車間 869次閱讀
- PIL的使用以及劃分圖像的皮膚區域 1670次閱讀
- 電子皮膚的應用_電子皮膚未來 9211次閱讀
- 詳解Xilinx公司Zynq? UltraScale+?MPSoC產品 3357次閱讀
- 電子皮膚研發歷史與電子皮膚的作用 7211次閱讀
- 基于Arm技術的16nm MPSoC開發套件Ultra96 6201次閱讀
- 一種新的基于電穿孔的皮膚高效核酸遞送方法 5187次閱讀
- 基于DSP的人體皮膚測量儀設計與實現方案[圖] 1271次閱讀
- 介紹了基于計算機視覺的方法檢測皮膚癌和白內障的方法 4289次閱讀
- 想成為PCB熟手,這96點你是必須要看的! 4460次閱讀
- 電子皮膚與主體交互有望取代智能可穿戴健康設備 2160次閱讀
下載排行
本周
- 1山景DSP芯片AP8248A2數據手冊
- 1.06 MB | 532次下載 | 免費
- 2RK3399完整板原理圖(支持平板,盒子VR)
- 3.28 MB | 339次下載 | 免費
- 3TC358743XBG評估板參考手冊
- 1.36 MB | 330次下載 | 免費
- 4DFM軟件使用教程
- 0.84 MB | 295次下載 | 免費
- 5元宇宙深度解析—未來的未來-風口還是泡沫
- 6.40 MB | 227次下載 | 免費
- 6迪文DGUS開發指南
- 31.67 MB | 194次下載 | 免費
- 7元宇宙底層硬件系列報告
- 13.42 MB | 182次下載 | 免費
- 8FP5207XR-G1中文應用手冊
- 1.09 MB | 178次下載 | 免費
本月
- 1OrCAD10.5下載OrCAD10.5中文版軟件
- 0.00 MB | 234315次下載 | 免費
- 2555集成電路應用800例(新編版)
- 0.00 MB | 33566次下載 | 免費
- 3接口電路圖大全
- 未知 | 30323次下載 | 免費
- 4開關電源設計實例指南
- 未知 | 21549次下載 | 免費
- 5電氣工程師手冊免費下載(新編第二版pdf電子書)
- 0.00 MB | 15349次下載 | 免費
- 6數字電路基礎pdf(下載)
- 未知 | 13750次下載 | 免費
- 7電子制作實例集錦 下載
- 未知 | 8113次下載 | 免費
- 8《LED驅動電路設計》 溫德爾著
- 0.00 MB | 6656次下載 | 免費
總榜
- 1matlab軟件下載入口
- 未知 | 935054次下載 | 免費
- 2protel99se軟件下載(可英文版轉中文版)
- 78.1 MB | 537798次下載 | 免費
- 3MATLAB 7.1 下載 (含軟件介紹)
- 未知 | 420027次下載 | 免費
- 4OrCAD10.5下載OrCAD10.5中文版軟件
- 0.00 MB | 234315次下載 | 免費
- 5Altium DXP2002下載入口
- 未知 | 233046次下載 | 免費
- 6電路仿真軟件multisim 10.0免費下載
- 340992 | 191187次下載 | 免費
- 7十天學會AVR單片機與C語言視頻教程 下載
- 158M | 183279次下載 | 免費
- 8proe5.0野火版下載(中文版免費下載)
- 未知 | 138040次下載 | 免費
 電子發燒友App
電子發燒友App

















 創作
創作 發文章
發文章 發帖
發帖  提問
提問  發資料
發資料 發視頻
發視頻
 上傳資料賺積分
上傳資料賺積分









評論