編輯
高光譜圖像分析
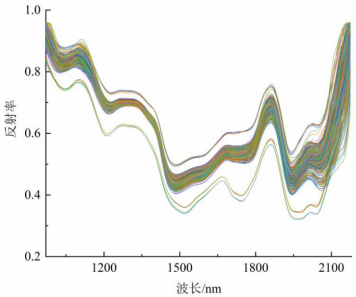
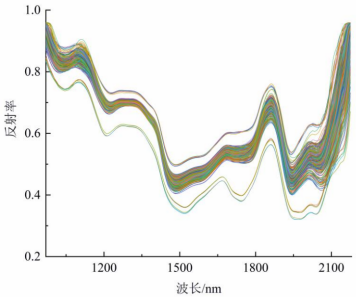
圖1為77個不同物質含量面粉樣品的原始平均光譜反射曲線。在901-2517nm波長范圍內,不同面粉的光譜具有相似的趨勢,但也存在一定差異,這些差異可能是面粉品種的內部化學成分和表面信息的差異造成的。但當波長在小于969nm和超過2174nm時由于掃描過程中能量過大,噪音等影響導致光譜曲線的變化趨勢不規則,因此選取969-2174nm波段的原始光譜數據進行后續工作。波長在969-1310、1470-1860、1935-2025和2040-2170nm處的顯著特征波峰和波谷與面粉中存在的蛋白質、淀粉和水分中的N-H、C-H、O-H的第一和第二泛音拉伸以及組合波段和彎曲振動有關。因此,利用NIR-HSI技術預測小麥粉中蛋白質、淀粉和水分含量是可行的。
編輯
圖1 面粉原始平均光譜反射曲線
樣本集劃分
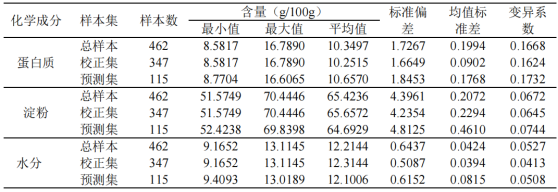
首先將面粉樣本劃分為校正集和預測集,然后進行多元數據分析。利用校正集樣品進行優化,建立定量模型。利用預測集樣本來證明最優化結果和所建立的定量模型的重復性能。采用KS算法按照3:1的比例劃分水分、蛋白質和淀粉的樣本集。使系統響應之間的歐氏距離最大化,均勻覆蓋多維空間。因此,選取347個樣本作為校正集,其余115個樣本作為預測集。校正集和預測集面粉樣品中蛋白質、淀粉和水分含量的分布如表3-1所示。校正集樣本包含了預測集樣本的變化范圍。這些數據表明,樣本集劃分方法的結果是合理的,所選擇的樣本構建模型具有較強的代表性。表3-1面粉中蛋白質、淀粉和水分含量的校正集和預測集的統計參考測量結果
編輯
面粉中蛋白質含量模型的建立
3.1 基于全波長的建模分析
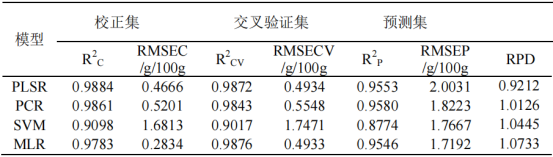
根據面粉高光譜圖像提取的全光譜數據及其對應的蛋白質含量參考值,建立全光譜校正模型,采用4種算法模型對蛋白質含量進行預測。表3-2給出了PLSR、PCR、SVMR和MLR相應優化校正模型的R2C、RMSEC、R2P、RMSEP、R2CV、RMSECV和RPD的結果。PCR為面粉蛋白質含量的最佳全波長預測模型。相應的R2C、R2CV和R2P分別為0.9861、0.9843和0.9580,相關的RMSEC、RMSECV和RMSEP分別為0.5201g/100g、0.5548g/100g和1.8223g/100g,RPD為1.0126。表3-2利用高光譜成像技術預測面粉蛋白質含量的模型性能
編輯
3.2 數據預處理
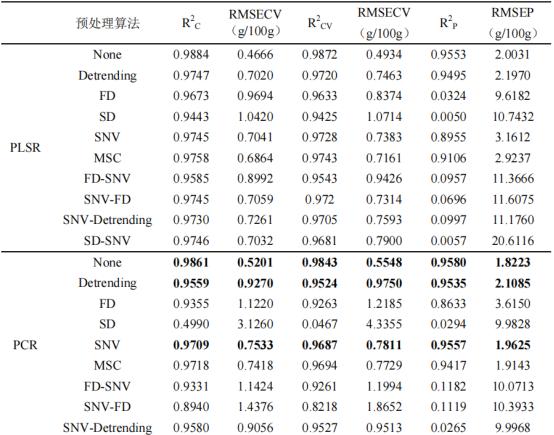
對原始光譜分別進行Detrending、FD、SD、SNV、MSC、FD-SNV、SNV-FD、SNV-Detrending和SD-SNV共9種預處理算法,預處理后的光譜數據分別建立PLSR、PCR、SVMR和MLR四類模型用于預測蛋白質含量。各預處理算法在Unscrambler中運行。各模型參數如下表3-3所示,結果表明,基于原始光譜的PCR模型對蛋白質含量的預測效果最好,PLSR、SVMR和MLR模型的預測效果略差于PCR模型,且SVMR模型存在一定程度的過擬合。PCR模型中9種預處理方法,Detrending和SNV的預測效果較好,其R2C、RMSEC、R2CV、RMSECV和R2P、RMSEP分別為0.9559和0.9709、0.9270g/100g和0.7533g/100g、0.9524和0.9687、0.9750g/100g和0.7811g/100g、0.9535和0.9557、2.1085g/100g和1.9625g/100g,但模型的效果和預測的精度均低于基于原始光譜的PCR模型,預處理效果不佳可能是由于一些關鍵信息失真。因此在后續工作中蛋白質含量預測模型的建立并未經過預處理。969-2174nm范圍內的光譜包含大量冗余信息,不利于提高模型的魯棒性和預測速度。因此,需原始光譜中選取特征波長進一步優化模型。表3-3基于不同預處理方法的PLSR、PCR、SVMR和MLR模型的蛋白質含量預測結果
編輯
3.3 提取特征波長
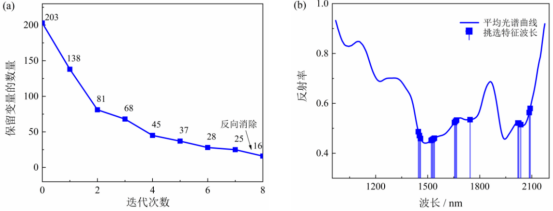
(1)基于IRIV算法提取面粉中蛋白質的特征波長IRIV是一種基于二進制矩陣變換濾波器(BMSF)的新型變量選擇方法。IRIV算法通過多次迭代剔除非信息變量和無關信息變量,保留有效信息變量。圖2(a)顯示了波長數隨迭代次數增加的變化過程。采用IRIV算法對蛋白質在962-2174nm波長范圍內共進行了8輪迭代。在前四輪迭代中,波長的數量從203急劇下降到45,因為許多無關信息波長被消除,然后在隨后的多輪迭代中緩慢下降。該結果在第7輪迭代時是穩定的,隨后反向消除了9個變量。從圖2(b)中可以看出,從原波長中選取的蛋白質特征波長數為16個(1452,1458,1464,1526,1532,1538,1544,1660,1666,1672,1750,2025,2030,2041,2090,2095nm),占總波長的7.88%。
編輯
圖2IRIV算法篩選面粉中蛋白質含量特征波長
(a:迭代次數剩余變量生長模式,b:挑選特征波長)
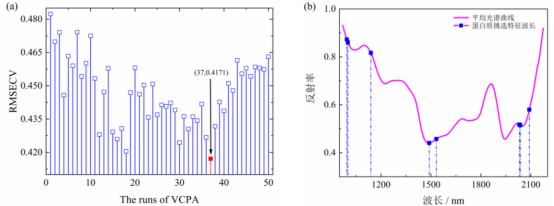
(2)基于VCPA算法提取面粉中蛋白質的特征波長VCPA基于指數遞減函數(EDF)和二進制矩陣采樣(BMS)迭代,選擇性能最優的特征波長子集。VCPA參數設置如下:EDF運行50次,BMS運行1000次,通過5倍交叉驗證確定所選波長,最優子集之比為0.1。圖3(a)為EDF運行過程中RMSECV的變化趨勢。隨著EDF的反復操作,特征空間縮小,RMSECV整體呈下降趨勢,當迭代次數為37次時,RMSECV最小為0.4171g/00g。最后選取RMSECV最小的變量子集,提取8個蛋白質含量的特征波長(994,1001,1139,1489,1532,2030,2036,2090nm)(圖3(b)),占總波長的3.94%。
編輯
圖3VCPA算法篩選面粉中蛋白質含量特征波長
a:選擇結果根據最小RMSECV確定特征波長;b:IRIV選擇的特征波長分布
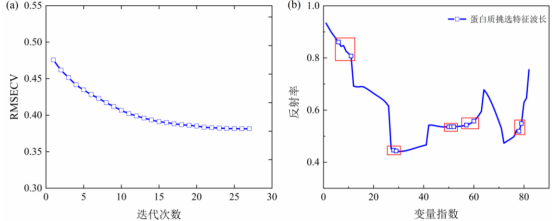
(3)基于IVISSA算法提取面粉中蛋白質的特征波長IVISSA結合全局搜索和局部搜索,以迭代方式智能優化光譜區間的位置、寬度和組合。圖4(a)為RMSECV在迭代過程中的變化趨勢。本研究中,在經過27次迭代后IVISSA算法篩選出蛋白質的82個特征波長。該算法提取了大量的特征波長且波段間距較小,一般來說,相似波長具有相同或相似的信息。因此,有必要進一步提取高光譜圖像降維的特征波長,以減少相鄰波段之間的無效信息,提高模型的運算速度。在IVISSA的基礎上,利用IRIV進一步選擇特征波長,并提出IVISSA-IRIV相結合,篩選出蛋白質的最佳特征波長數為11個(1001,1145,1470,1477,1732,1738,1744,1773,1791,2030,2079nm)(圖4(b)),占總波長的5.42%。
編輯
圖4IVISSA和IVISSA-IRIV算法篩選面粉中蛋白質含量特征波長
(a:RMSECV在迭代過程中的變化趨勢;b:所選特征波長的序號)
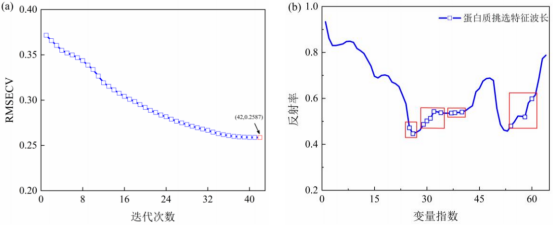
(4)基于MASS算法提取面粉中蛋白質的特征波長MASS算法主要采用連續模型空間收縮和加權迭代策略來獲得模型空間中的高性能模型。在此過程中,MASS應用隨機樣本程序,結合離群值掩蔽和變量組合效應,得到最優模型。圖5(a)為迭代過程中RMSECV的變化趨勢,為下降趨勢。MASS算法經過42次迭代后,RMSECV最小值降至0.2587g/100g,蛋白質保留64個特征波長。本文將MASS算法與IRIV算法相結合,建立了一種混合變量選擇方法來解決特征變量問題。MASS-IRIV算法最終得到的變量子集如圖5(b)所示。從64個變量集中,提取了13個蛋白質含量的特征波長(1452,1470,1612,1630,1642,1684,1708,1744,1756,1767,1969,2023,2095nm),占總波長的6.40%。
編輯
圖5MASS和MASS-IRIV算法篩選面粉中蛋白質含量特征波長
(a:RMSECV在迭代過程中的變化趨勢;b:所選特征波長的序號)
(5)基于IRF算法提取面粉中蛋白質的特征波長IRF是一種基于隨機蛙PLS框架的新型波長選擇方法。在此過程中,IRF計算300次迭代生成的300個變量子集中每個波長的選擇概率,并按降序排列。對每組波長分別進行交叉驗證,得到RMSECV。RMSECV最小組中的波長即為所選波長。如圖6(a)所示,選取前81個變量子集作為蛋白質的特征波長。IRF最終選擇了976-1019、1101-1164、1415-1489、1507-1550、1581-1593、1618-1678、1690-1773、1785-1797、1873-1907和1964-2101nm共105個特征波長[圖6(b)]。IRF保留了許多波長變量,結合IRIV進一步選取IRF的運行結果,以提高模型的魯棒性和運算速度。蛋白質的波長數從105個減少到20個(1007,1013,1151,1158,1164,1433,1439,1446,1660,1666,1702,1708,1714,1750,1791,1797,2030,2036,2074,2095nm),其中有效減少的光譜維數如圖6(c)所示,占全波長的9.85%。
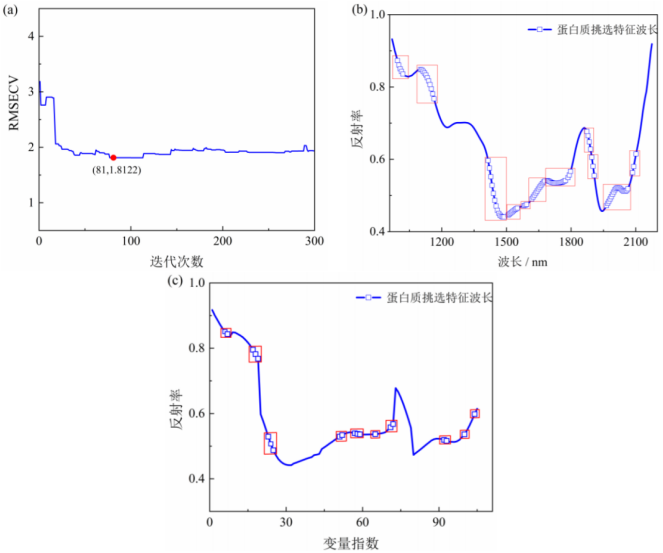
編輯
(a:RMSECV在迭代過程中的變化趨勢;b:IRF算法所選特征波長的序號;c:IRF-IRIV算法所選特征波長的序號)
3.4 最優建模效果的比較
首先評估了基于全波段近紅外高光譜數據建立的PLSR、PCR、SVMR和MLR模型的性能。合適的特征波長提取算法有利于模型獲得更好的魯棒性和準確性,反之則可能會對模型的準確性和穩定性造成破壞,降低預測精度[100]。將全波段波長和提取的特征波長作為PCR模型的輸入數據,評估特征波長提取對預測模型的影響。不同模型對蛋白質含量的預測結果如圖7(a)所示。對比所建預測模型的預測性能,基于特征波長的模型對面粉蛋白質含量均能獲得較好的預測效果。雖然IVISSA、MASS和IRF算法選擇的特征波長數較多,提高了模型的性能,但模型的簡化效果并不明顯。因此,應進一步結合預測性能較好的IRIV算法提取特征波長。在蛋白質含量的定量分析模型中,基于全波長PCR模型的預測模型效果中R2P=0.9580,RMSEP=1.8223g/100g,RPD=1.0126。在蛋白質含量的預測模型中,對所選擇的特征波長提取算法進行了評價和比較,驗證了特征波長提取算法的準確性和有效性。最優模型IVISSA-IRIV-PCR預測蛋白質含量,提取了11個特征波長,其中R2C=0.9883,R2P=0.9859,RMSEC=0.4769g/100g,RMSEP=1.1580g/100g,RPD=1.5935。圖7(b)是基于IVISSA-IRIV-PCR模型對面粉中蛋白質含量的預測值和實際值的散點圖。虛線表示蛋白質實際值與預測值之間理想相關性的回歸線。樣本點在回歸線附近分布緊密,說明模型的預測性能較好。在本研究中,面粉根據面筋含量可分為低筋面粉和高筋面粉。高筋面粉的蛋白質含量較高,淀粉含量較低,而低筋面粉的淀粉含量較高,蛋白質含量較低。因此,樣品的蛋白質含量分布在兩個簇中是合理的。本研究選取高筋面粉和低筋面粉作為樣品,是為了擴大模型的檢測范圍,為今后模型的應用奠定基礎。綜上所述,特征波長的選擇可以降低高光譜數據的高維度和復雜性,提高預測模型的精度和計算速度。結果表明,近紅外高光譜成像技術可以準確地實現面粉中蛋白質含量的檢測。綜上所述,近紅外高光譜成像技術是一種適用于面粉化學成分檢測的方法。
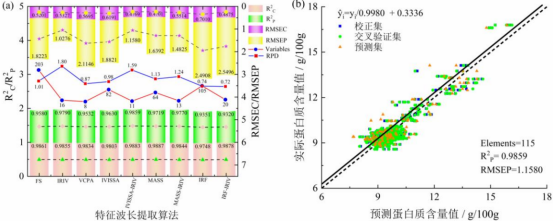
編輯
圖7a:基于全光譜和挑選特征光譜的蛋白質含量的PCR模型的預測結果;
b:IVISSA-IRIV-PCR模型獲得的預測蛋白質含量的散點圖
3.5 面粉中蛋白質含量的可視化分布
與傳統光譜技術相比近紅外高光譜成像技術可以同時提供樣品的光譜和圖像信息[1011。采用線性色標對提取的特征波長建立的蛋白質模型進行像素級處理可視化的偽彩色圖,其中紅色區域表示蛋白質含量高,紫色區域代表其含量低,如圖8所示。利用IVISSA-IRIV提取的11個特征波長,建立了預測高光譜圖像上每個像素蛋白質含量的簡化模型。最后構建檢測指標可視化圖(圖8)。預測樣品的顏色變化自動集中在一個線性色條上,其中不同的顏色對應著面粉中蛋白質含量的不同值。可視化圖可以直觀地反映不同品種樣品甚至同一品種樣品中蛋白質含量的空間變化,有利于掌握物質含量的相對分布。偽彩色圖可以顯示不同面粉中基本化學成分的分布情況方便食品加工企業和采集者直觀地選擇需要的面粉。
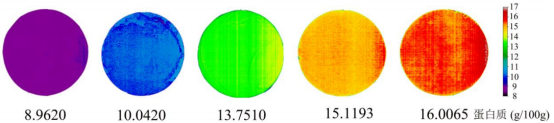
編輯
圖8 面粉中蛋白質含量的可視化圖
推薦:
便攜式高光譜成像系統 iSpecHyper-VS1000
專門用于公安刑偵、物證鑒定、醫學醫療、精準農業、礦物地質勘探等領域的最新產品,主要優勢具有體積小、幀率高、高光譜分辨率高、高像質等性價比特點采用了透射光柵內推掃原理高光譜成像,系統集成高性能數據采集與分析處理系統,高速USB3.0接口傳輸,全靶面高成像質量光學設計,物鏡接口為標準C-Mount,可根據用戶需求更換物鏡。
審核編輯 黃宇
-
成像
+關注
關注
2文章
243瀏覽量
30545 -
高光譜
+關注
關注
0文章
345瀏覽量
9999
發布評論請先 登錄
相關推薦
高光譜成像系統:光譜成像技術在海域目標探測中的應用
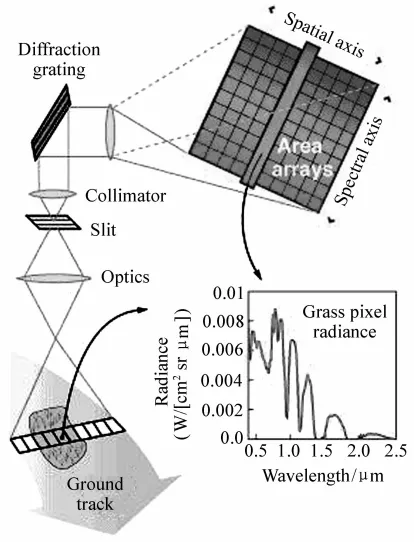




 基于高光譜成像技術檢測面粉中的蛋白質含量
基于高光譜成像技術檢測面粉中的蛋白質含量













評論