大豆食心蟲成蟲的蟲卵會附著于大豆表面,孵化的幼蟲會啃食大豆,對大豆產量和品質造成嚴重影響。這種現象在我國各大豆產區普遍發生,如發現和預防得不及時會使得大豆產量嚴重下降。在大豆病害的檢測上,人工可視化調查作為實踐中最基本的直接方法,至今仍在使用。然而,這種方法需要相關植物表型和植物病理學的專業知識;另一種常見的植物病害檢測技術可以稱為生物分子法,但生物分子技術需要詳細的取樣和復雜的處理方法,與人工調查方法相比,這些方法更具專業性和周期性。這兩種技術具有基本性、有效性,但總是需要手動去檢測,導致復雜的工作和較大的勞動量。
近年來,深度學習技術在植物病害分類中的成功應用,為大豆病害的研究提供了新思路,該技術通過將卷積網絡與高光譜結合,能夠對目標進行有效分類。高光譜技術可視為光譜學的一部分,它可以從多個光譜帶中獲取光譜信息,一些有效波段對病害引起的大豆細微變化具有很高的敏感性,從而可以區分不同的病害類型。卷積網絡是一種特殊的深度學習技術,在圖像處理和提取特征方面具有突出的能力。
1實驗部分
1.1樣本制備
測試集中所需要食心蟲大豆樣本由專業農業機構提供,將只成蟲放入大豆中使其于大豆上產卵,5d后采集附著蟲卵的大豆,10d后采集附著食心蟲幼蟲的大豆,30d后采集被啃食的大豆。分別對正常的大豆以及上述三種大豆拍攝高光譜圖像,每類樣本數量為20。
1.2高光譜成像系統
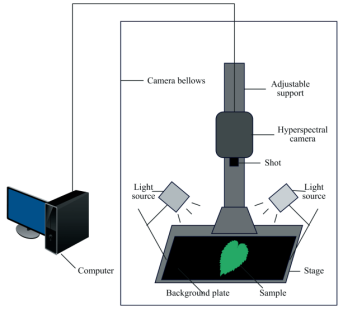
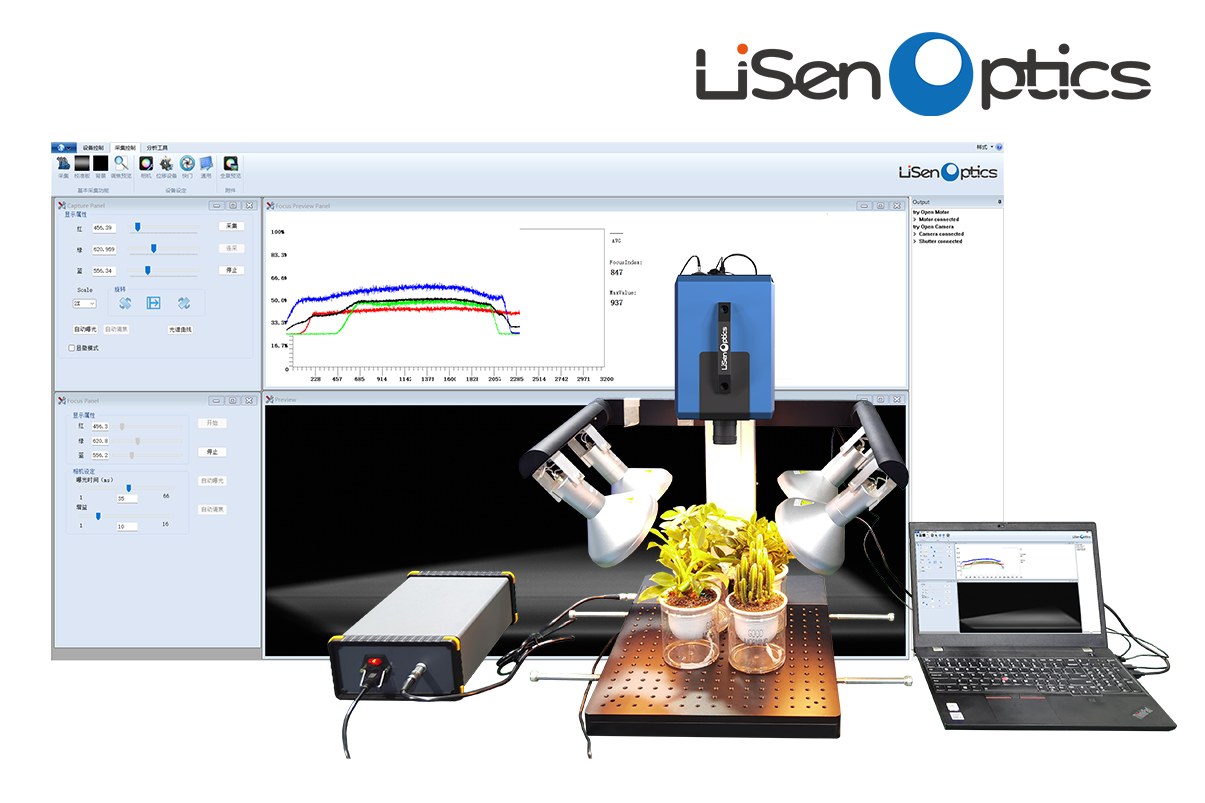
由軟件進行圖像采集,高光譜成像系統的組成部分有電控平移臺,4盞功率為150w的鹵素燈,CCD相機,一臺計算機五個部分,采集的圖像包含256個光譜波段。它們的光譜范圍為383.70~1032.70nm,整個采集過程在暗箱中完成,避免了環境光的影響,如圖1所示。

圖1系統結構圖
1.3高光譜圖像采集
采集圖像時,曝光時間為18ms,平臺的移動速度設置為1.50cm.s-1,鹵素燈與平臺之間的夾角設置為50°。先采集白板圖像W和暗背景圖像B,然后對大豆樣本進行圖像采集,得到256個波段的高光譜圖像。采集到的大豆樣本合成的RGB圖如圖2所示
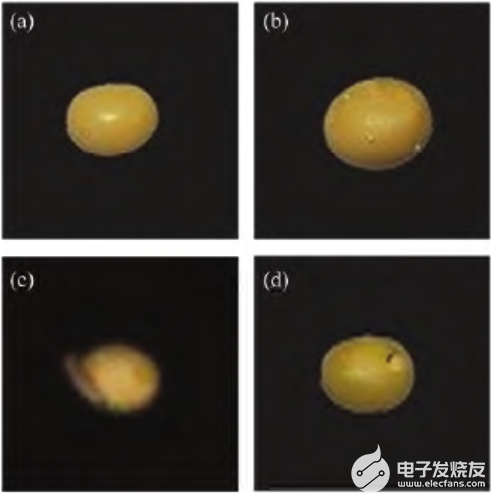
圖2大豆樣本高光譜圖像
1.4數據預處理
采集圖像過程中為了緩解可能出現的漫反射,樣本不均勻,基線漂移等問題,對采集圖像進行了Savitzky-Golay濾波處理,針對光照不均勻的問題,使用了黑白校正的預處理方法。高光譜圖像的波段數量太多,許多波段的數據是冗余的;為了避免這些冗余數據造成的影響,使用了主成分分析法進行降維處理,選取前30個高光譜波段作為特征波段。以樣本為中心截取了50*50像素的正方形區域,使得單個高光譜樣本數據的大小為50*50*30。
1.5圖像檢索模型原理
3D-R-D模型的內容主要包括一個用于提取特征的3D卷積網絡,一個用于幫助網絡產生有效特征的DCH損失函數。其中網絡模型為3D-Resnet18,該預訓練模型的原始數據集包括Kinetics-700(K)和MomentsinTime(M)兩個數據集,前者一共700個類,每個類包括超過600個來自You-Tube的人類動作視頻,后者是包括100萬個視頻的數據集,使用這樣的預訓練模型,可以得到較好的初始化參數。預訓練模型并不能直接使用,在網絡結構的改變上,本工作僅僅去除最后的分類層,并添加一個從高維映射到低維的hash層,這樣不僅使用了模型中的初始化參數,還能利用網絡訓練來進行降維。在損失函數的設計上,為得到效果優良的相似度特征,文獻中經常使用成對損失函數來更新這些特征提取算法的可學習參數。專家學者通過設計合理的損失函數,使得每一對輸入樣本如果相似就讓它們的特征相互靠近,不相似的樣本對特征距離相互遠離,充分利用了樣本標簽之間的相似性;部分學者對輸入圖像的監督信息進行編碼,對輸出特征進行正則化,以逼近所需的離散值,該方法設計了一個閾值m,當不相似對的特征距離大于m時不提供損失貢獻。部分學者設計了一個基于柯西分布的成對損失,它對漢明距離大于給定漢明半徑閾值的相似圖像對造成了顯著的懲罰,也充分利用了標簽信息。將中的損失函數與交叉熵函數進行結合,使得最后的特征信息包含分類損失和標簽之間的相似度信息。通過引入一個哈達碼矩陣,改進交叉熵損失,通過添加分類中心,增強了分類損失的特征信息。在特征距離的計算上,由于最后的特征都是低維的二進制碼,所以使用漢明距離進行衡量,并且由于維度較低,不同樣本之間特征距離的計算速度極快。
2結果與討論
2.1模型使用與分析
如上所述。本工作使用預訓練模型,并將分類層改為hash層,損失函數使用的是DCH,訓練集為CAVE,iCVL和NUS,測試集為采集的大豆樣本,訓練時用于微調以適應應用場景的訓練集則和文獻中描述的一樣,就是使用光譜數據集CAVE,iCVL和NUS作為數據集。CAVE是一個包含32個場景的數據集,iCVL高光譜數據集由計算機視覺會議收集,包含農村,城市,植物,公園,室內這些場景,NUS數據集則包含了一些普通場景和水果,而上述的四種類別的大豆數據則作為分類實驗中的測試集和檢索集。利用圖像檢索進行分類的步驟如下所示:
(1)利用訓練集訓練得到一個網絡模型,這個模型能夠對輸入的同一類高光譜樣本輸出相似的二進制特征。
(2)從大豆樣本的每一類中隨機取出10個樣本組成檢索集,對檢索集提取特征并儲存到文件A中,剩下的大豆樣本作為測試集。
(3)模擬新采集樣本的分類:將測試集中每一個樣本依次提取特征,并用漢明距離與文件A中的數據進行相似度匹配排序,從檢索集取出的排名前5個樣本中相同標簽最多的即為這個測試樣本的類別。
(4)實驗得到的準確率即為正確的測試個數占總測試樣本數量的比重,重復上述步驟,反復實驗取平均值。在最近的大豆食心蟲分類研究中,文獻使用了小樣本分類,采用了MN,MAML,3D-RN的方法,也解決了高光譜圖像數據樣本不足的問題,它的實驗結果如表1所示。
表1不同分類模型在4-way5-shot情況下的檢測結果
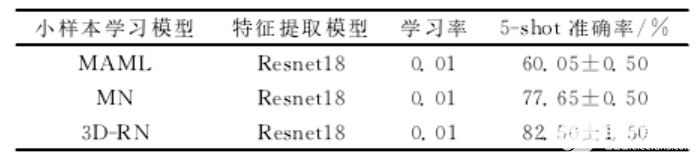
由此發現,使用Resnet18的網絡結構,具有良好的效果,為了研究圖像檢索進行分類的有效性,也為了能和文獻進行對比,通過使用不同的預模型,結合不同損失函數,設計了如表2實驗。
表2不同損失函數下的檢索性能
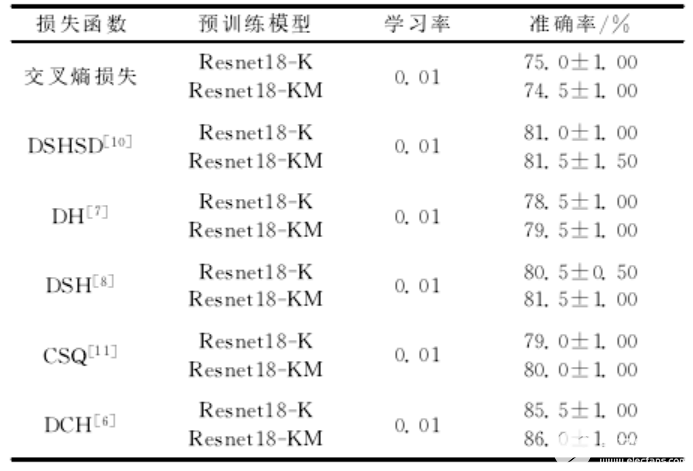
在表2中,Resnet18-K表示使用了Kinetics-700(K)為數據集的預訓練模型,Resnet18-KM,表示該預訓練模型還使用了MomentsinTime(M)數據集,從實驗結果中發現,盡管使用了更多的數據,本實驗在性能上,有所提升但是比較細微;和原來的交叉熵損失函數相比,DH使用成對損失提高了5%的分類準確率,在使用M閾值之后,DSH又有所提高,DSHSD盡管結合分類損失和成對損失,但是性能和DSH幾乎沒有區別,由此可見,單純的分類損失并不適用于本次實驗,CSQ引入了中心損失,在改善交叉熵后,確實提高了5%左右的準確率,和DSH差不多,而DCH在使用柯西分布后,達到了86%的準確率,相比于之前效果最好的3D-RN實驗,提高的準確率。
3結論
近年來,使用高光譜成像對農業病蟲害檢測已經應用地十分廣泛,但是樣本數量少的問題仍然需要解決,本文通過采集不同時期的樣本,利用樣本之間的相似度信息,構建了一種基于圖像檢索的分類方法,在模型上,從視頻檢索中得到啟發,利用大量數據訓練的3D預訓練網絡,獲得了較好的初始化參數,利用3DCNN,使得不同波段間數據的相似性能夠被利用起來,通過對不同損失函數的比較,DCH利用柯西分布,能較好地提取到樣本之間地相似信息,它的準確率達到了86.0±1.00,從而解決了實際問題,這是一種新穎的高光譜檢測方法,為高光譜檢測的相關研究提供了一種新的思路。
審核編輯 黃昊宇
-
光譜檢測
+關注
關注
0文章
12瀏覽量
6671 -
高光譜
+關注
關注
0文章
345瀏覽量
10000
發布評論請先 登錄
相關推薦
高光譜相機:農業監測革命新利器!
做病蟲害監測,要如何選擇合適的地物光譜儀廠家
Spectricity攜手高通為智能手機提供光譜圖像傳感器成像技術





 基于圖像檢索的大豆食心蟲蟲害高光譜檢測
基于圖像檢索的大豆食心蟲蟲害高光譜檢測













評論